Un software ayudará a buscar fármacos para 20 enfermedades tropicales desatendidas
El desarrollo, liderado por científicos de la Fundación Instituto Leloir (FIL) y del Instituto de Investigaciones Biotecnológicas "Rodolfo Ugalde" (IIBIO), orienta sobre posibles blancos terapéuticos y drogas activas contra patógenos que causan tuberculosis, la enfermedad de Chagas, malaria y lepra, entre otras patologías.
 Los directores del estudio, Fernán Agüero (Izq.) y Ariel Chernomoretz, y el primer autor del trabajo, Lionel Urán Landaburu.
Los directores del estudio, Fernán Agüero (Izq.) y Ariel Chernomoretz, y el primer autor del trabajo, Lionel Urán Landaburu.Las enfermedades tropicales desatendidas, como Chagas, malaria y lepra, afectan de manera desproporcionada a 1.500 millones de personas en los países de bajos ingresos y en desarrollo, donde son la principal causa de años de vida perdidos por discapacidad y muerte prematura. Por su falta de atractivo para la industria farmacéutica, la mayoría se trata con viejos medicamentos que tienen varias décadas de uso, a menudo con resultados deficientes.
Ahora, los investigadores de la FIL y del IIBIO desarrollaron “TDR Targets 6”, la última versión de una herramienta bioinformática que permite, de manera accesible, buscar información sobre posibles blancos terapéuticos en genomas completos de patógenos que causan veinte de estas enfermedades, lo que podría orientar el hallazgo o desarrollo de fármacos más efectivos y seguros.
“Nuestra herramienta pretende facilitar la búsqueda de estrategias terapéuticas para enfermedades desatendidas”, señaló Ariel Chernomoretz, codirector del desarrollo y jefe del Laboratorio de Biología de Sistemas Integrativa de la FIL.
El software está organizado en dos secciones principales: blancos terapéuticos derivados de genes y proteínas de más de 20 patógenos que causan tuberculosis (Mycobacterium tuberculosis), la enfermedad de Chagas (Trypanosoma cruzi), malaria (Plasmodium falciparum), toxoplasmosis (Toxoplasma gondii), y lepra (Mycobacterium leprae), entre otras patologías.
La herramienta también contiene información de 5.300 blancos terapéuticos validados y más de 2 millones de compuestos bioactivos para seleccionar aquellos que pudieran tener un efecto terapéutico.
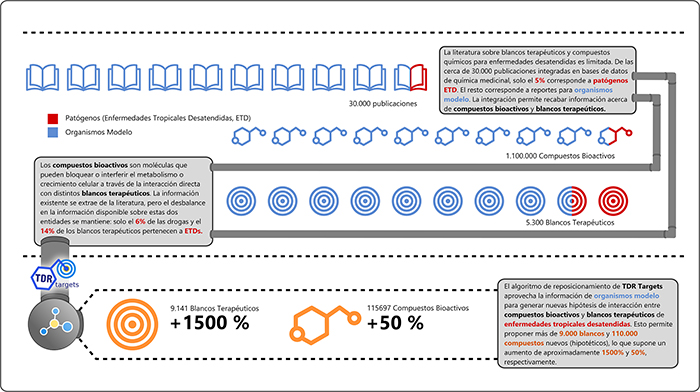 Infografía que muestra el funcionamiento de la herramienta bioinformática TDR Targets. El algoritmo busca aprovechar la información existente para enfermedades y organismos en donde existe mucha inversión en investigación y desarrollo para identificar potenciales nuevos blancos terapéuticos y compuestos químicos para enfermedades menos estudiadas (desatendidas). Créditos: Lionel Urán Landaburu.
Infografía que muestra el funcionamiento de la herramienta bioinformática TDR Targets. El algoritmo busca aprovechar la información existente para enfermedades y organismos en donde existe mucha inversión en investigación y desarrollo para identificar potenciales nuevos blancos terapéuticos y compuestos químicos para enfermedades menos estudiadas (desatendidas). Créditos: Lionel Urán Landaburu.El software puede ser útil para el “reposicionamiento de drogas”, esto es, la indicación para nuevos usos de medicamentos ya aprobados para otras condiciones.
“Como sus perfiles de seguridad son conocidos, el reposicionamiento facilita una aplicación más rápida”, subrayó Chernomoretz, también investigador del CONICET y profesor del Departamento de Física de la Facultad de Ciencias Exactas y Naturales de la UBA.
Reducir el tiempo y costo del desarrollo de medicamentos es algo especialmente importante para este tipo de enfermedades, en las que el financiamiento es escaso.
El estudio también fue dirigido por Fernán Agüero, investigador del CONICET y jefe del Laboratorio de Genómica y Bioinformática del Instituto de Investigaciones Biotecnológicas "Rodolfo Ugalde" (IIBIO), que depende de la UNSAM; y el primer autor es Lionel Uran Landaburu, becario doctoral del CONICET en el laboratorio de Agüero.
Otros investigadores argentinos que participaron del estudio, publicado en la revista “Nucleic Acids Research”, son Ariel Berenstein, actualmente en el Instituto Multidisciplinario de Investigaciones en Patologías Pediátricas (IMIPP), que depende del CONICET y funciona en el Hospital de Niños “Ricardo Gutiérrez”; y Santiago Videla, exbecario posdoctoral de la FIL y del CONICET.
El estudio contó con el apoyo de GlaxoSmithKline Argentina y la Agencia Nacional de Promoción de la Investigación, el Desarrollo Tecnológico y la Innovación (Agencia I+D+i) y forma parte de un Proyecto Bilateral de Cooperación Indo-Argentina con financiamiento conjunto del Departamento de Ciencia y Tecnología de la India (DST) y el Ministerio de Ciencia y Tecnología de Argentina (MINCyT).
